Verbundprojekt: DINaMid |
| Genom-basierte Findung neuer antimikrobieller Naturstoffe in mikrofluidischen Chips |
| Laufzeit: 01.10.09-30.09.12 |
| Verbundkoordinator: Dr. Markus Nett, Hans-Knöll-Institut Jena (HKI), Jena |
| Projektpartner: (alphabetische Reihenfolge) |
| Dr. Thomas Henkel, Institut für Photonische Technologien e.V. |
| Dr. Markus Nett, Hans-Knöll-Institut Jena (HKI) |
| Dr. Martin Roth, Hans-Knöll-Institut Jena (HKI) |
| Vor dem Hintergrund der zunehmenden Ausbreitung antibiotika-resistenter Krankheitserreger kommt der Suche nach neuen antimikrobiellen Wirkstoffen groÃe Bedeutung zu. Naturstoffe sind aufgrund ihrer strukturellen Diversität und ihrer optimierten Affinität zu biologischen Zielstrukturen ideale Kandidaten für die Wirkstoffsuche. Die bioinformatische Analyse von mikrobiellen Genomen belegt, dass deren Potential zur Produktion von Naturstoffen bei weitem nicht ausgeschöpft ist. Diese Erkenntnis hat in den vergangenen Jahren wesentlich zu einer Renaissance der Naturstoff-Forschung beigetragen. Ãber sog. âGenome Miningâ-Strategien ist es heute möglich, gezielt nach strukturell neuartigen Metaboliten zu suchen und diese zu isolieren. |
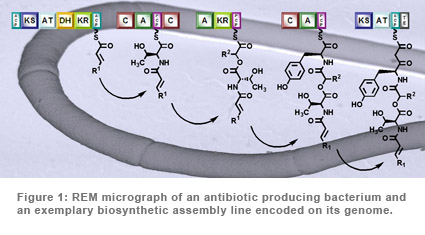 |
| Ein neues Konzept, welches wir innerhalb des GenoMik-Transfer-Programms evaluieren, ist die genom-basierte Wirkstoffsuche in einem mikrofluidischen System. Viele Biosynthesewege unterliegen einer komplexen Regulation, die von äuÃeren Faktoren beeinflusst werden kann. Durch die Ãnderung einzelner Kultivierungsparameter lassen sich ruhende Gencluster aktivieren und auf diese Weise metabolische Ãnderungen induzieren. Die Miniaturisierung eines solchen Prozesses in einem Chip erlaubt hohe Durchsatzraten und ist damit auch für industrielle Anwendungen attraktiv. Zu diesem Zweck werden zunächst separierte Flüssig/flüssig-Kompartimente generiert, die mit dem zu untersuchenden Bakterium beimpft werden. Es folgt eine Inkubationsphase, in welcher eine Nährstoff-abhängige und damit Kompartiment-spezifische Wirkstoffbildung stattfindet. Für die Detektion werden GFP überexprimierende Reporterkeime zudosiert und das Wachstum derselbigen über Fluoreszenzmessungen bestimmt. Die strukturelle Charakterisierung neuer Naturstoffe erfolgt nach einem Scale-up über spektroskopische Methoden und wird durch bioinformatische Vorhersagen unterstützt. |
 |
Referenzen: |
| 2. Nett, M., Ikeda, H., Moore, B. S. (2009) Genomic basis for natural product biosynthetic diversity in the actinomycetes. Nat. Prod. Rep. 26, 1362-1384. |
| 1. Martin, K., Henkel, T., Baier, V., Grodrian, A., Schön, T, Roth, M., Köhler, J. M., Metze, J. (2003) Generation of larger numbers of separated microbial populations by cultivation in segmented-flow microdevices. Lab Chip 3, 202-207. |
GenoMik-Transfer - Anwendungsorientierte Genomforschung an Mikroorganismen
Neue Broschüre: Facetten der Genomforschung
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)



