Verbundprojekt: IPG |
| Integrierte Phänotyp-/Genotyp-Plattform zur Identifizierung verborgener genetischer Targets und Targetkombinationen |
| Laufzeit: 01.12.10-30.11.13 |
| Verbundkoordinator: Stephan Hans, Evonik Industries AG, Halle/Westfalen |
| Projektpartner: (alphabetische Reihenfolge) |
| Dr. Christopher Bauser, GATC Biotech GmbH |
| Dr. Hans Peter Fischer, Genedata Bioinformatik GmbH |
| Stephan Hans, Evonik Industries AG |
| Dr. Jörn Kalinowski, Universität Bielefeld |
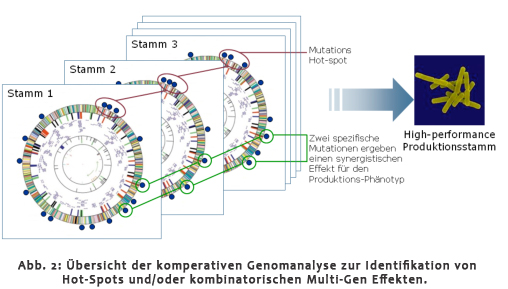
| Vor über einem Jahrzehnt wurde bereits die Bedeutung der Genomsequenzen von industriell relevanten Organismen erkannt [1]. Nachdem zuerst die Genomsequenzen der Typstämme aufgeklärt wurden, folgte die Sequenzierung der Produktionsstämme. Diese zeigten oft mehrere hundert Mutationen im Vergleich zur Typstamm-Sequenz. Nach heutigem Stand der Technik existiert keine Methode, mit der die für den Produktionsphänotyp relevanten Mutationen identifiziert und von dem komplexen Hintergrund der Produktionsphänotyp-irrelevanten oder sogar -nachteiligen Mutationen unterschieden werden können. Die "Integrierte Phänotyp-Genotyp" (IPG) Plattform versucht, für dieses Problem eine methodische Lösung zu finden. Um dieses Ziel zu erreichen, wird eine Hochdurchsatz-Plattform aufgebaut und auf genetisch unterschiedliche C. glutamicum-Stammgenealogien angewendet. Wir gehen davon aus, dass für die statistisch abgesicherte Identifikation von komplexen Mutationsereignissen eine Vielzahl valider Genotyp- und Phänotypdatensätze, basierend auf verschiedenen Produktionsstamm-Mutanten, erforderlich ist. Alle Daten werden im Rahmen des Projektes erhoben und benutzt, um systematisch und statistisch fundiert kombinierte produktions-relevante Mutationsprofile in komplexen genetischen Hintergründen zu identifizieren und zu charakterisieren. |
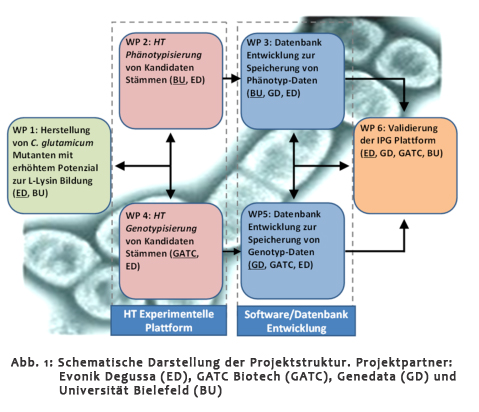 |
| Die Projektziele sind: i) Genotyp- und Phänotypinformationen zu integrieren, ii) im GroÃmaÃstab Stammvergleiche auf Genotyp- und Phänotyp-Basis durchzuführen, iii) "Hot-Spots" von Mutationen zu identifizieren und iv) innovative Targets zur Eliminierung metabolischer "bottlenecks" aufzuzeigen. |
 |
Referenzen: |
| [1] Kalinowski J, et al. , J Biotechnol. 2003 Sep 4;104(1-3):5-25. |
GenoMik-Transfer - Anwendungsorientierte Genomforschung an Mikroorganismen
Neue Broschüre: Facetten der Genomforschung
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)



