Verbundprojekt: PathControl |
| Unterdrückung des pflanzenpathogenen Pilzes Rhizoctonia solani durch das pflanzenwachstumsfördernde Bakterium Bacillus amyloliquefaciens in der pflanzlichen Wurzelzone |
| Laufzeit: 01.01.10-30.06.13 |
| Verbundkoordinator: Prof. Dr. Rainer Borriss, ABiTEP GmbH, Berlin |
| Projektpartner: (alphabetische Reihenfolge) |
| Prof. Dr. Rainer Borriss, ABiTEP GmbH |
| Prof. Dr. Anton Hartmann, Helmholtz Zentrum München |
| Prof. Dr. Alfred Pühler, Universität Bielefeld |
| Dr. Andreas Schlüter, Universität Bielefeld |
| PD Dr. Philippe Schmitt-Kopplin, Helmholtz Zentrum München |
| Industriepartner: |
| ABiTEP GmbH, Berlin |
| Durchführung einer molekularen Analyse der genetischen Faktoren, die die Fähigkeit des pflanzen-wachstums-fördernden Bakteriums Bacillus amyloliquefaciens FZB42 [1,2] zur Kolonisierung von Pflanzenwurzeln (Salat) in Gegenwart des phytopathogenen Pilzes Rhizoctonia solani bestimmen. |
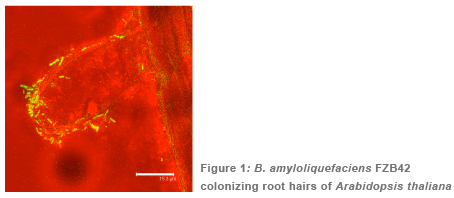 |
| Die Expression der Gene, die die Fähigkeit von B. amyloliquefaciens zur Kolonisierung der Rhizosphäre determinieren, wird auf Transkriptions- (RNA-Microarray) und Translationsebene (Proteomics) untersucht. Die antagonistische Wechselwirkung zwischen phytopathogenem Pilz und Bacillus und vice versa ist dabei von besonderem Interesse. Besonderes Augenmerk wird auf den direkten Nachweis der Bildung von Sekundärmetaboliten durch B. amyloliquefaciens im Bereich der pflanzlichen Wurzelzone gelegt. Grundlage für die Entwicklung eines effizienten Biokontrollpräparates auf der Basis von FZB42 ist die Beantwortung der Frage, ob die Biokontrollwirkung von FZB42 überwiegend auf (i) der Produktion antifungaler Sekundärmetaboliten, (ii) auf einer direkten Wachstumskompetition während Kolonisierung der Pflanzenwurzeln, oder (iii) auf eine Verstärkung der induzierten, systemischen Resistenz der Pflanze gegenüber pathogenen Mikroorganismen zurückgeht. |
 |
Referenzen: |
| 2. Borriss et al. (2011) Relationship of Bacillus amyloliquefaciens clades associated with strains DSM7T and FZB42: a proposal for Bacillus amyloliquefaciens subsp. amyloliquefaciens subsp. nov. and Bacillus amyloliquefaciens subsp. plantarum subsp. nov. based on their discriminating complete genome sequences. Int J Syst Evol Microbiol. 61(8):1786-1801. |
| 1. Chen et al. (2007) Comparative analysis of the complete genome sequence of the plant growth-promoting bacterium Bacillus amyloliquefaciens FZB42. Nature Biotechnol. 25(9):1007-14. |
GenoMik-Transfer - Anwendungsorientierte Genomforschung an Mikroorganismen
Neue Broschüre: Facetten der Genomforschung
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)
Wissenschaftler entschlüsseln die Baupläne des Lebens (pdf; 12,3 Mb)



